[Publicidad]
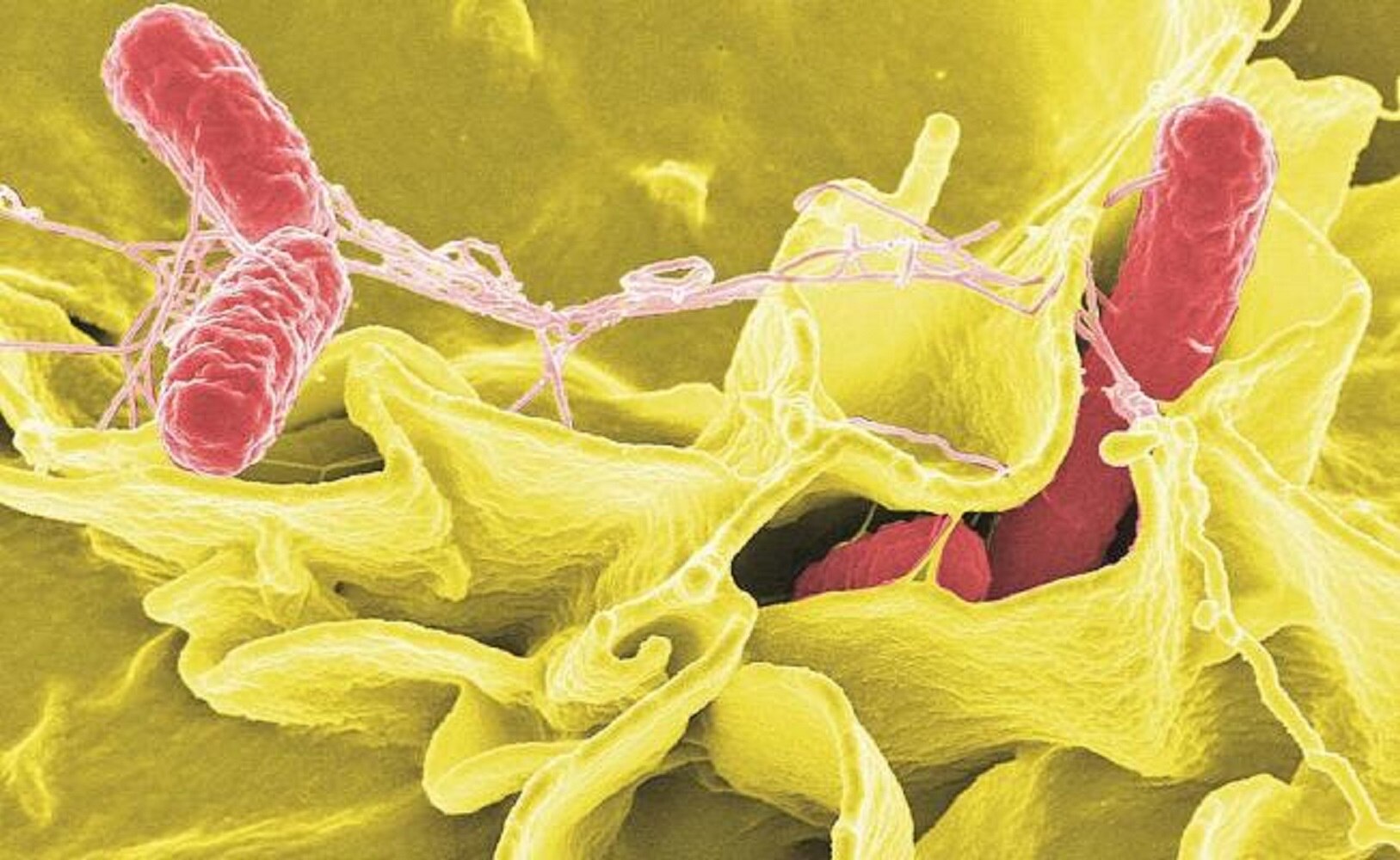
La resistencia bacteriana a los antibióticos es un problema de salud pública mundial. En 2008, las cepas de Staphylococcus aureus resistentes a los antibióticos ocasionaron más muertes en Estados Unidos que el virus de inmunodeficiencia humana (VIH). Y en México, más de 70% de las infecciones causadas por Escherichia coli corresponden a cepas resistentes a la penicilina, la ampicilina, la tetraciclina y la combinación trimetoprima-sulfametoxazol.
En septiembre de 2016, la Asamblea General de la ONU se comprometió a luchar contra este problema porque se calcula que en 2050 habrán muerto más de 10 millones de personas por infecciones bacterianas que no responden a los antibióticos.
“Las bacterias se vuelven resistentes a los antibióticos debido a su altísima capacidad de cambio y adaptación que les permite vivir en ambientes extremos (muy ácidos, calientes, fríos o salinos) a los que otras formas de vida no podrían ni acercarse”, dice Maximino Aldana González, investigador del Instituto de Ciencias Físicas y del Centro de Ciencias de la Complejidad (C3) de la UNAM.
Por un lado, la tasa de reproducción de las bacterias es muy alta (E. coli, por ejemplo, se reproduce cada 20 minutos en condiciones favorables, y su crecimiento es exponencial); y por el otro, al reproducirse experimentan mutaciones en su material genético, y algunas de estas mutaciones producen cambios a partir de los cuales pueden adaptarse mejor a condiciones ambientales adversas, como un medio con antibióticos.
“Con tantas bacterias reproduciéndose no es extraño que surjan algunos individuos que por pura variabilidad genética sean más resistentes a los antibióticos. Y son ellos los que dan lugar a poblaciones de bacterias altamente resistentes a estos fármacos”, apunta el investigador.
Uno de los más importantes descubrimientos en biología consistió en saber que las características de un organismo no sólo están determinadas por la información escrita en sus genes, sino también por la rapidez con que dicha información se lee. Esto significa que esos mismos genes pueden producir características fenotípicas distintas, dependiendo de la rapidez con que se lea la información contenida en ellos.
“Esa rapidez (o nivel de actividad) depende de factores ambientales como la temperatura, la acidez, la salinidad o los antibióticos. Por lo tanto, estos factores pueden determinar cuáles características fenotípicas se desarrollarán y cuáles no, lo que daría origen a organismos diferentes, incluso si éstos tienen los mismos genes.”
Los procesos que cambian la actividad de los genes sin cambiar su información se conocen como procesos epigenéticos.
Sistema MDR
Algunas bacterias, como E. coli y Salmonella, contienen un grupo de genes que al activarse dan lugar a unas bombas de proteínas (bombas de eflujo) que se anclan en la membrana celular. La función de estas bombas es expulsar hacia el exterior de la bacteria el líquido que se encuentra en su interior, esto es, en el citoplasma.
“Dicho grupo de genes recibe el nombre de sistema Multi Drug Resistance (MDR, por sus siglas en inglés). Cuando no hay un antibiótico, este sistema está desactivado. Y se activa por medio de procesos epigenéticos inducidos por el antibiótico, que cambian el nivel de actividad de los genes que lo conforman, sin cambiar su información genética”, indica Aldana González.
Al activarse el sistema MDR se empiezan a producir las bombas de eflujo, que expulsan de la bacteria el antibiótico que está entrando, con lo que se evita que la concentración de éste dentro de la célula bacteriana llegue a niveles letales. El sistema MDR puede considerarse un primer mecanismo de defensa reversible, no específico y hereditario, al que recurren muchas bacterias gram negativas cuando son atacadas con un antibiótico. Es no específico porque las bombas expulsan no sólo el antibiótico que ataca a la célula, sino también nutrientes y otras sustancias que la bacteria necesita para su desarrollo. A la bacteria no le conviene tener estas bombas funcionando todo el tiempo. Por lo tanto, cuando el antibiótico es eliminado del medio, el sistema MDR se apaga. En este sentido es reversible.
“Entender los procesos epigenéticos y la manera como afectan el fenotipo de los organismos vivos es, desde mi punto de vista, uno de los grandes retos de la biología de sistemas al que se enfrentarán las nuevas generaciones de científicos. Sólo así podremos ganar la guerra a las bacterias patógenas”.
Resistencia reversible
Había varias preguntas sin respuesta. Una de ellas era: ¿qué genera la diversidad en una población de bacterias? Porque si una población de bacterias es atacada con un antibiótico, algunas morirán pero otras no. Esto significa que no todas son iguales, aunque vengan de la misma “madre”.
Ahora bien, si algunas mueren y otras no, entonces no todas son idénticas. ¿Qué las hace diferentes? Aldana González y sus colegas observaron que, una vez que se les quita el antibiótico, las que sobreviven apagan sus bombas de eflujo, pero no inmediatamente, sino después de algunas generaciones.
“En colaboración con un equipo de la Universidad de Harvard, aquí, en la UNAM, desarrollamos un modelo matemático del sistema MDR que nos permite entender claramente cuál es la diferencia a nivel molecular entre las bacterias que mueren y las que sobreviven”, asegura el investigador.
Las bombas de eflujo tardan cierto tiempo en encenderse. Si se aplica un antibiótico en una población de bacterias, muchas morirán y otras, las que pudieron encender esas bombas lo suficientemente rápido, sobrevivirán.
Patrones de metilación del ADN
Al preguntarse cuál era la diferencia a nivel molecular entre las bacterias que no pudieron encender las bombas de eflujo a tiempo y las que sí lo hicieron, los investigadores universitarios encontraron que tenía que ver con los patrones de metilación del ácido desoxirribonucleico (ADN).
La transcriptasa es una molécula que se mueve a lo largo del ADN, leyendo la información que contiene éste y produciendo ácido ribonucleico (ARN), que a su vez genera las proteínas y las bombas de eflujo. Pero hay otra molécula —la metilasa— que va poniendo obstáculos en el ADN. Esos obstáculos se conocen como patrones de metilación. Al moverse por el ADN, la transcriptasa los va encontrando y, dependiendo de la estructura del patrón de metilación, algunas veces la metilasa se ve obligada a detenerse; esto hace que lea más lentamente la información genética para producir las bombas de eflujo.
“Estos obstáculos en el ADN no son parte de la información genética. Los patrones de metilación son grupos metilo que se van poniendo en la hebra del ADN. Si bien se conocen como patrones de metilación, los obstáculos se van poniendo al azar.”
Según sea el patrón de metilación, la transcriptasa va a leer más o menos rápido la información genética. Esa rapidez o lentitud en la lectura es la diferencia fundamental entre las bacterias que sobreviven y las que mueren.
“Por puro azar, algunas moléculas tienen mejores patrones de metilación que otras para producir bombas de eflujo con más rapidez. Aunque después vienen otros mecanismos de defensa para que se enciendan estas bombas, el patrón de metilación es fundamental, y esto lo demostramos”, comenta Aldana González.
Lo que se debe intentar ahora es cambiar los patrones de metilación de las bacterias y poner patrones de metilación adecuados para que la transcriptasa tenga tantos obstáculos que no pueda generar las bombas de eflujo a tiempo.
“Eso les toca a los microbiólogos y biotecnólogos. Ellos pueden producir las mutaciones para que los genes que generan la metilasa hagan más lenta a la transcriptasa. Con nuestro modelo teórico les señalamos esta diferencia fundamental.”
Diversidad bacteriana
Es un hecho que si el patrón de metilación siempre fuera el mismo, las bacterias no sobrevivirían. Para que las poblaciones de bacterias sobrevivan, debe haber diferencias entre ellas. Si todas fueran iguales, se podría poner una concentración de antibiótico suficientemente alta para que las matara a todas; sin embargo, precisamente por esa diversidad, ningún antibiótico acaba con todas. Los patrones de metilación puestos al azar son los que generan la diversidad bacteriana inicial. Después, las células sobrevivientes podrán generar diversidad permanente mediante mutaciones en el ADN. Pero la diversidad producida por los patrones de metilación es fundamental al inicio para que algunas bacterias puedan sobrevivir al antibiótico.
“Basta con que una bacteria tenga los patrones de metilación adecuados para generar las bombas de eflujo lo suficientemente rápido y así esté en condiciones de luchar contra el antibiótico empleado. Este hallazgo fue nuestra primera contribución al estudio de la resistencia bacteriana”, afirma Aldana González.
Reversibilidad
La reversibilidad también está basada en la diferencia aleatoria en los patrones de metilación, porque no todas las bacterias tienen los mismos. En una población de bacterias a las que se aplica un antibiótico, la mayor parte muere pero sobreviven dos o tres que tienen los patrones de metilación adecuados y que empiezan a generar bombas de eflujo rápidamente. Las hijas de estas bacterias tendrán las bombas de eflujo encendidas y heredarán los patrones de metilación que les permitirán seguir produciendo otras.
“A medida que esta población crece, hay variaciones en los patrones de metilación de las células hijas. En cada generación aparecen bacterias con patrones favorables y otras con patrones desfavorables. En presencia de un antibiótico, los patrones desfavorables generan bombas de eflujo lentamente, mientras que los patrones favorables las generan más rápidamente.”
Pero cuando ya no hay antibiótico se intercambian los papeles: los patrones de metilación que producen bombas de eflujo rápidamente ahora son desfavorables porque están expulsando nutrientes, y esto hace que las bacterias ya no puedan crecer tan rápido.
“Por su parte, las bacterias con patrones de metilación que producen bombas de eflujo lentamente son las que tienen mayor ventaja reproductiva en ausencia del antibiótico. Después de 10 ó 20 generaciones bajan totalmente su actividad, pero no porque hayan apagado los genes, sino por los patrones de metilación que hacen que la transcriptasa vaya leyendo cada vez más lentamente la información genética hasta que se llega a un punto en que una población ya no tiene las bombas de eflujo encendidas porque ya no hay nada que las ataque. Ésta es nuestra principal aportación a la investigación de la resistencia bacteriana”, finaliza el investigador.
[Publicidad]
Más información
Tendencias
Cómo denunciar si te hicieron stickers en tu trabajo

Vida Q
“Welcome to Derry”; las diferencias y similitudes entre la precuela de HBO y la novela “It” de Stephen King

Tendencias
¿Qué insectos se comen en Querétaro y cómo se preparan?

Tendencias
¿Qué hay arriba de la pirámide del Pueblito en Querétaro?